清華新聞網11月8日電 近日,清華大學藥學院田博學課題組研究提出了一個基于蛋白質語言模型和對比學習的蛋白質-小分子結合位點預測模型(CLAPE-SMB),并整理了蛋白質-小分子結合位點數(shù)據集UniProtSMB,發(fā)現(xiàn)該模型在UniProtSMB的測試集上達到了0.699的MCC,優(yōu)于其他模型。與基于結構的預測方法相比,CLAPE-SMB特別適用于缺乏精確實驗結構的蛋白質,如固有無序蛋白(IDP)。這就為理解蛋白質-小分子相互作用提供新的視角,并為藥物設計提供新的可能。
蛋白質與小分子的相互作用在很多生命活動中發(fā)揮關鍵作用,例如催化反應、信號傳導、代謝調控等。小分子通過與蛋白質的特定位點結合,調控其活性,進而影響生物體內的多種功能。為了找到蛋白質上的這些結合位點,科學家們采用了許多實驗方法,如表面等離子體共振(SPR)、質譜分析和X射線晶體學等高分辨率成像技術。這些實驗方法能精準定位結合位點,但耗時且資源需求大。計算方法為結合位點的預測提供了高效的補充,主要分為基于蛋白質結構和基于序列的預測模型。基于結構的模型,如ScanNet和DeepSite,利用蛋白質的三維結構信息來確定潛在的結合位點,精度較高,但對蛋白質結構的依賴限制了預測范圍、增加了應用難度。相比之下,基于序列的模型擺脫對結構的依賴,使用簡單但精確度較低。如GraphBind和DeepProSite,在僅使用序列信息的情況下表現(xiàn)一般。因此,目前基于蛋白質序列的小分子結合位點的預測仍然是一個具有挑戰(zhàn)性的問題。
為了解決目前模型準確度低的問題,田博學課題組提出了CLAPE-SMB。CLAPE-SMB由三個模塊組成。第一是蛋白質語言模型ESM-2,將一維的序列信息編碼為二維的特征矩陣。第二是多層感知機(MLP),將二維信息轉化為殘基級別的小分子結合概率。第三是損失函數(shù)模塊,包括解決類別不平衡的focal loss和對比學習損失函數(shù)(TCL)。
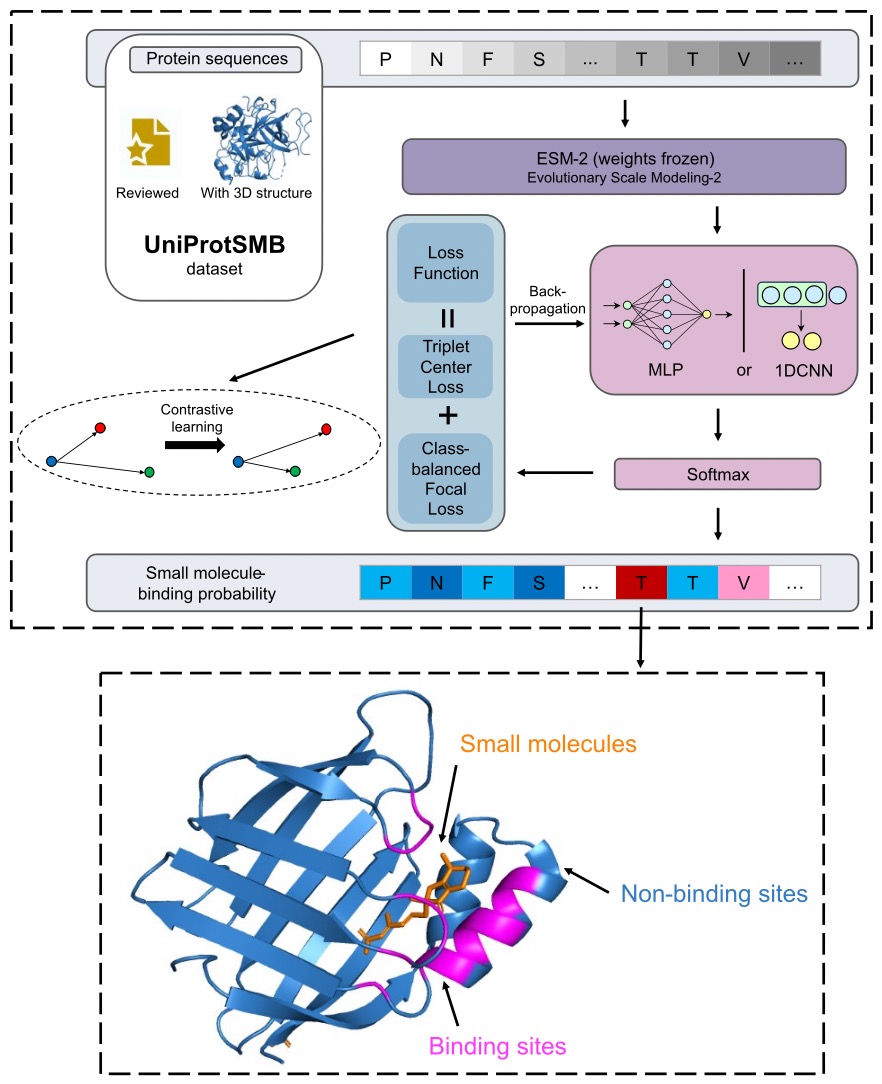
圖1.CLAPE-SMB的結構
為了使CLAPE-SMB有更好的泛化能力,田博學課題組構建了UniProtSMB數(shù)據集作為訓練集。研究人員從UniProtKB數(shù)據庫中篩選出具有三維結構和小分子結合位點的實驗驗證蛋白質,去除長度過長的蛋白質并標注標簽。隨后,進行聚類去除冗余蛋白,形成UniProtSMB數(shù)據集。最后,將UniProtSMB數(shù)據集劃分為訓練集、驗證集和測試集。
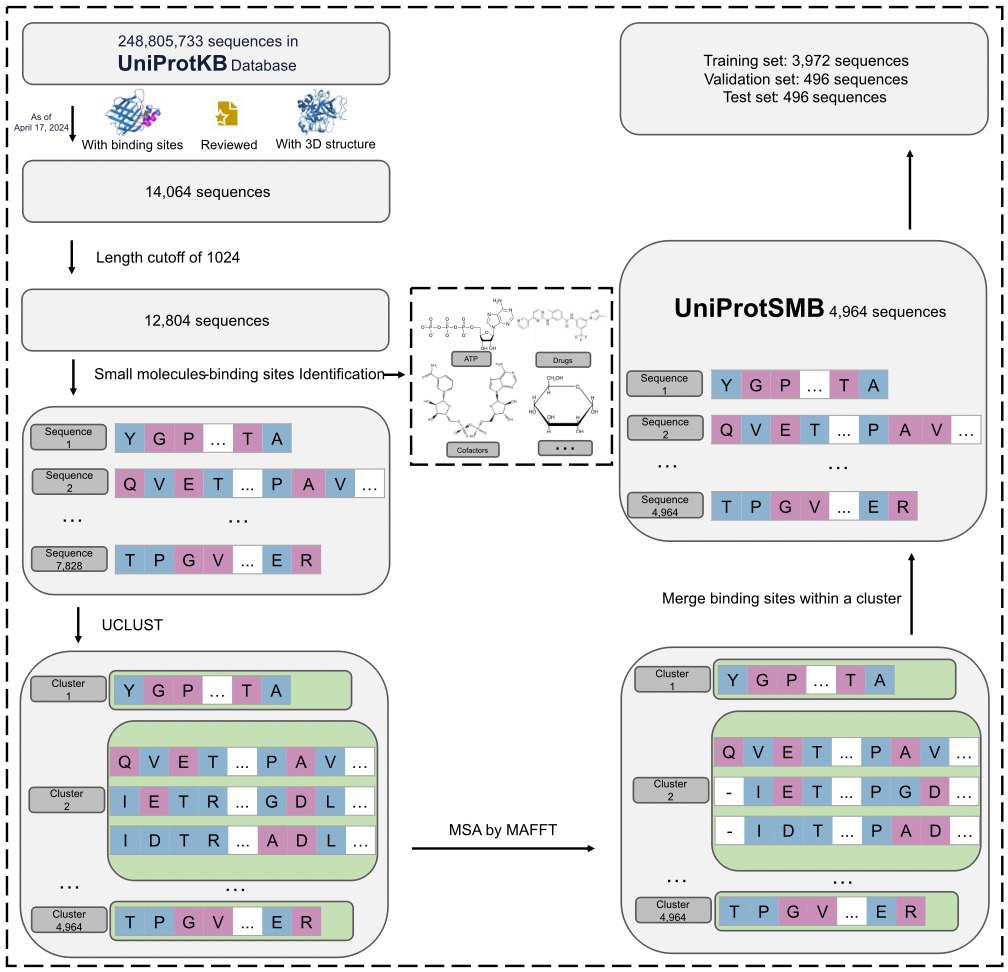
圖2.UniProtSMB數(shù)據集的構建流程
IDP沒有穩(wěn)定的三維結構,因此依賴準確結構信息進行預測的模型很難處理IDP。研究人員構建了IDP數(shù)據集,CLAPE-SMB在其上獲得了0.815的MCC。進一步,研究人員選取了兩個在固有無序區(qū)域(IDR)上有小分子結合位點的IDP進行案例研究,結果顯示CLAPE-SMB能夠準確預測出IDR上的結合位點。
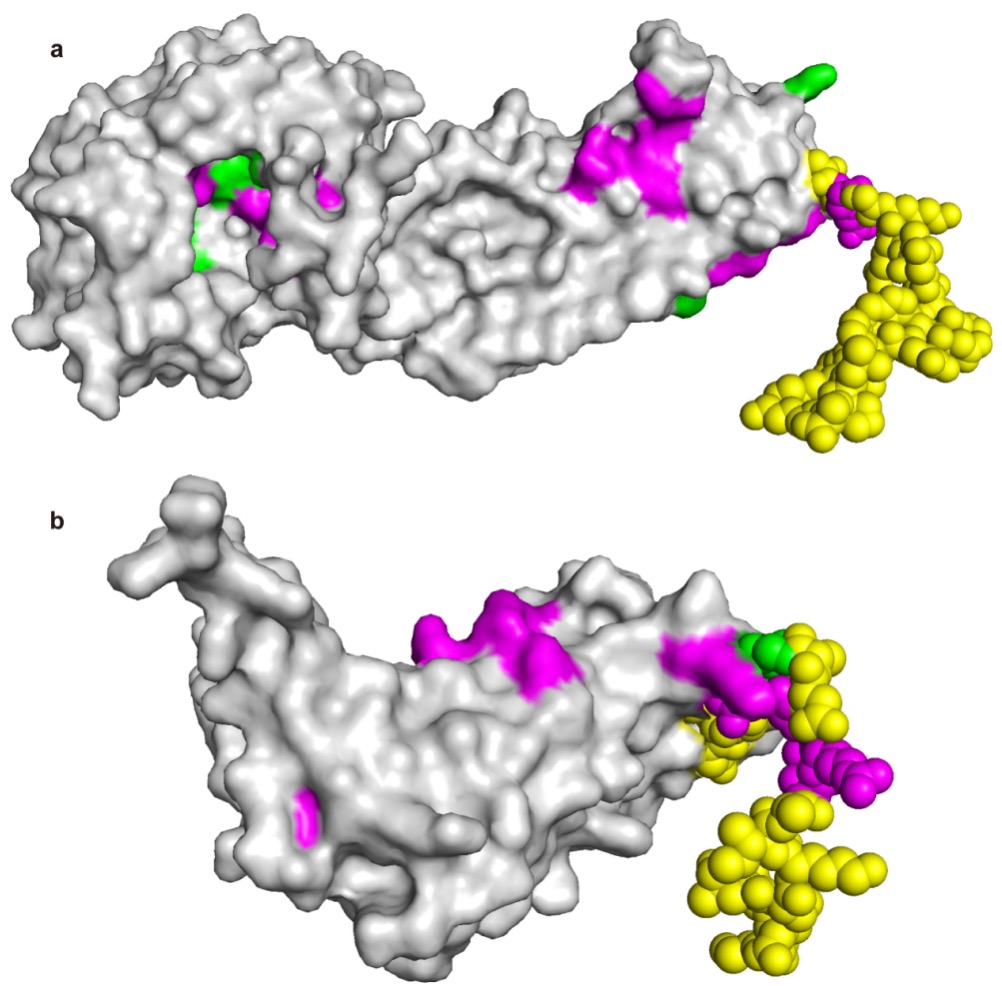
圖3.固有無序蛋白的案例研究
絕大多數(shù)蛋白質尚未解析結構,其中包括大量膜蛋白,而膜蛋白通常與多種小分子結合。未來,CLAPE-SMB可以用于預測膜蛋白的小分子結合情況。此外,CLAPE-SMB還能夠結合特定小分子的SMILES信息,僅預測該小分子是否與某蛋白存在結合位點,并定位具體的結合殘基。這一功能有望應用于虛擬篩選和老藥新用等領域。
該研究提出了CLAPE-SMB,它將預訓練的蛋白語言模型與對比學習相結合,實現(xiàn)了高精度的小分子結合位點預測,尤其適用于沒有晶體結構的蛋白質。研究人員在基于sc-PDB、JOINED和COACH420構建的非冗余SJC數(shù)據集上對CLAPE-SMB進行了訓練和測試,獲得了0.529的MCC。此外,基于UniProtKB數(shù)據構建的UniProtSMB數(shù)據集上,CLAPE-SMB在測試集上達到了0.699的MCC。對于包含336條非冗余序列的固有無序蛋白數(shù)據集,CLAPE-SMB的MCC高達0.815。對DAPK1、RebH和Nep1的案例分析進一步證明了該工具在藥物設計中的潛在應用價值。
相關研究成果以“基于預訓練蛋白質語言模型和對比學習的蛋白質-小分子結合位點預測”(Protein-small molecule binding site prediction based on a pre-trained protein language model with contrastive learning)為題,于11月6日發(fā)表于《化學信息學雜志》(Journal of Cheminformatics)。
清華大學藥學院副教授田博學為論文通訊作者,藥學院本科生王玨和田博學課題組已畢業(yè)博士生劉宇帆為論文共同第一作者。課題得到北京生物結構前沿研究中心、清華大學篤實專項、清華大學-北京大學生命科學中心的支持。
論文鏈接:
https://doi.org/10.1186/s13321-024-00920-2
供稿:藥學院
題圖設計:趙存存
編輯:李華山
審核:郭玲
